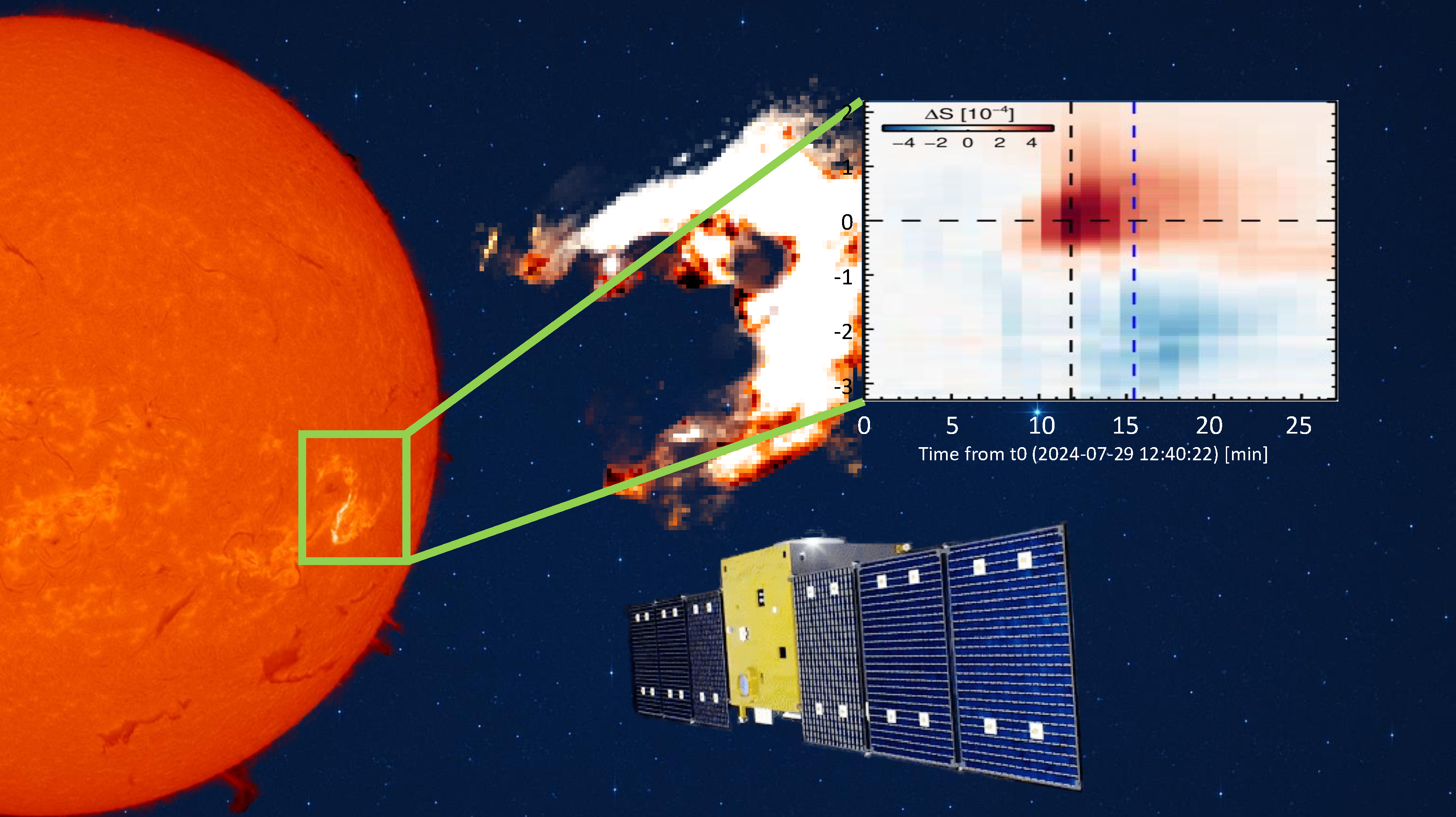
科技日报记者 马爱平
近日,中国农业科学院深圳农业基因组研究所(岭南现代农业科学与技术广东省实验室深圳分中心)刘永鑫研究员团队推出全新开源工具EasyAmplicon第2版,新版本在保留短读扩增子分析功能基础上,全面支持第三代全长扩增子测序数据分析,并优化了可视化功能,为微生物分类和定量研究提供更简单、易用的分析环境,现已免费开放使用。相关研究成果发表在《先进科学》上。
扩增子测序是解析微生物群落多样性的关键技术。传统短读测序仅覆盖基因片段,分辨率有限。长读长测序技术可获取全长基因序列,可提供高分辨率,但缺乏标准化分析流程。

为此,研究团队开发了适用于全长微生物组扩增子测序的分析流程,EasyAmplicon 2.0可实现自动化分析,涵盖质控、去噪、分类注释等步骤,支持SILVA、GTDB等主流数据库,并可生成30余种可视化图表。测试显示,在双核处理器环境下,其2小时内即可完成2万条读长的样本分析,适用于从探索性研究到大规模队列的全流程需求。
(中国农业科学院深圳农业基因组研究所供图)




 网友评论
网友评论