科技日报记者 罗云鹏
6月20日,中国科学院深圳先进技术研究院定量合成生物学全国重点实验室、合成生物学研究所合成微生物组学研究中心马迎飞团队在国际学术期刊《微生物组》发表最新研究成果,首次系统揭示小鼠、猪、食蟹猴三种模式动物肠道噬菌体的组成特征与分布规律。
噬菌体占肠道病毒的90%以上,作为肠道菌群的关键调控因子,与炎症性肠病、结直肠癌、糖尿病等疾病密切相关。
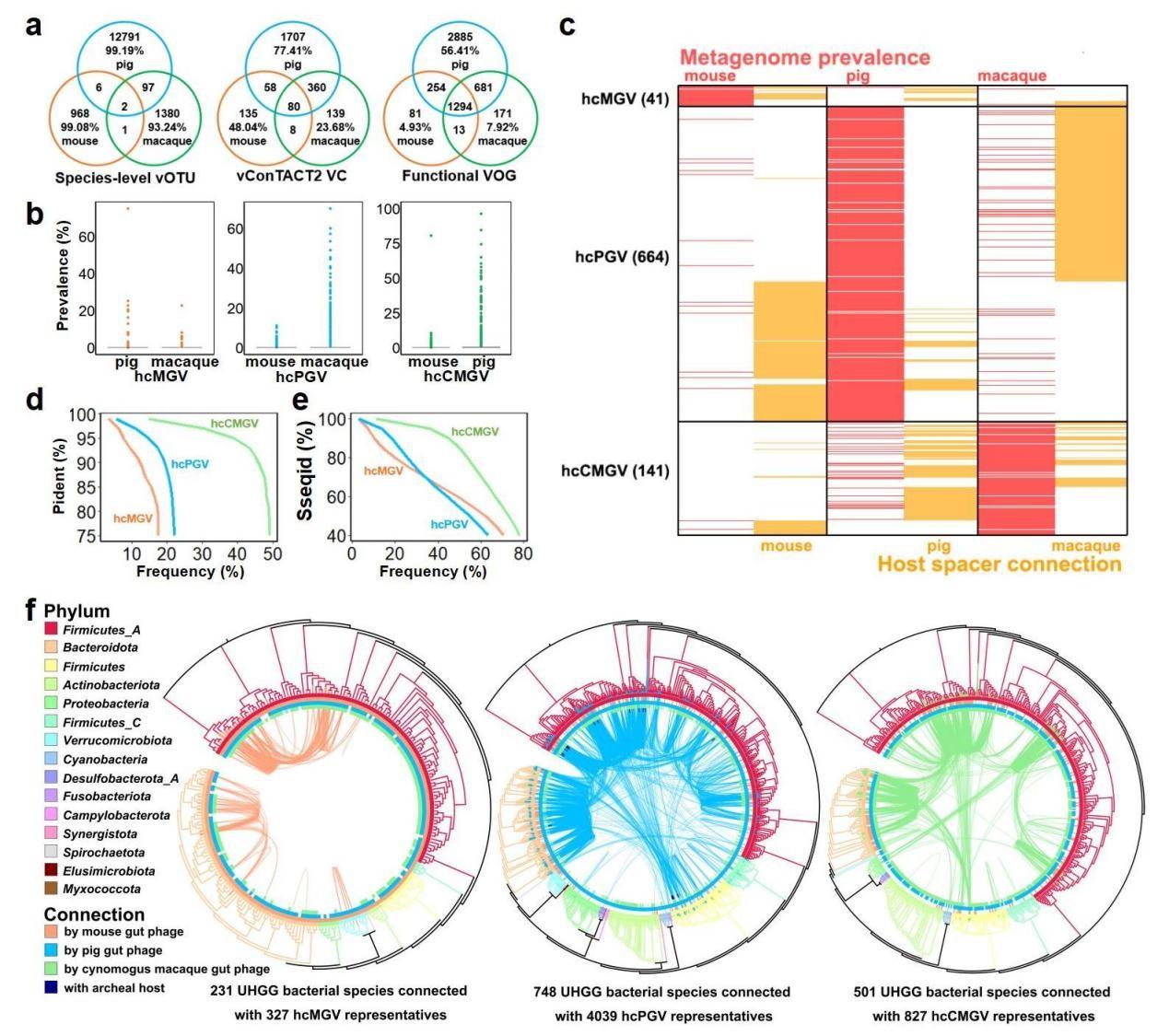
研究团队利用生物信息学方法,对约1500个小鼠、猪和食蟹猴肠道宏基因组进行高效标准化分析,成功构建了三种模式动物的高质量数据库,其中包括977条小鼠、12896条猪以及1480条食蟹猴种水平、完整度超过90%的高质量病毒序列。
经比对发现,大部分病毒序列属于有尾噬菌体,显著扩展了模式动物肠道噬菌体多样性。
基于基因组编码的蛋白相似性,研究团队在上述病毒序列中鉴定出71个分布率超过60%的高分布病毒群。其中,分布率最高的5个病毒群与国际病毒分类委员会已知分类病毒关联性较弱,但与其他物种的高分布病毒群存在关联,提示模式动物肠道噬菌体可能存在跨物种传播。
研究团队在三组数据库中共鉴定出315个crAss样噬菌体,其中182个与肠肚噬菌体科亲缘关系较近,部分形成独立于已知属的新分支。
此外,研究团队还在肠道数据库中鉴定出245条基因组长度超过200kb的巨型噬菌体,部分携带CRISPR-Cas系统。

值得一提的是,这些噬菌体编码的Cas14蛋白结构完整,最小的仅含153个氨基酸,具备开发新型基因编辑工具的潜力。
通过序列比对分析,研究团队进一步揭示这些巨型噬菌体的CRISPR间隔序列可匹配细菌、其他噬菌体甚至自身基因组片段,表明其CRISPR-Cas系统具有广泛的潜在功能多样性。
为探究模式动物与人类肠道微生物的关联性,研究团队通过核酸和蛋白水平比对发现,食蟹猴的肠道微生物组与人类的相似性高于小鼠和猪。
而在CRISPR spacer匹配分析中,食蟹猴数据库的匹配成功率超过50%,进一步证实了食蟹猴是研究人类肠道微生物更理想的动物模型。
马迎飞表示:“未来,该研究的范围或可拓展至肠道RNA病毒领域,探索噬菌体的功能机制及其与宿主的相互作用,有望在肠道病毒组的医学应用领域取得更多突破性进展。”
(研究团队供图)